¿Qué es Linked Open Data?
Antes de pasar a detallar las cuatro bases de datos definiremos brevemente el concepto de Linked Open Data. Este término surge de la web semántica, como parte o desarrollo de ésta.
Se trata de un método para publicar datos estructurados de tal forma que pueda ser enlazado de forma interna y por lo tanto ser más útil. Surge de la idea de que hay información disponible que no está accesible con los actuales métodos de búsqueda.
El científico de la computación Tim Berners Lee estableció este concepto, resaltando la necesidad de utilizar URIs (Enlaces que no varían con el tiempo, y por lo tanto son duraderos), aprovechar la tecnología Http para localizar la información, e incluir enlaces a otras URI relacionadas de forma que se potencie el descubrimiento de información en red.
Fig 1. Representación de los recursos Linked Open Data presentes en Internet. Los enlaces permiten llegar a cualquier parte de esta "nube". Puede ver la imagen a mayor tamaño en este enlace.
Para que un recurso cumpla con el Linked Open Data debe...
- Estar en la web: Utiliza el protocolo Http, no es necesario crear un nuevo protocolo o usar algo más complicado.
- Constituir datos que una máquina pueda procesar.
- Ser un formato no propietario.
- Cumplir los estándares RDF (Modelo de datos para metadatos)
- Incluir enlaces RDF utilizando los mencionados enlaces URI.
Podrás encontrar más información sobre Linked Open Data al final del Bibliotema, en la sección de enlaces.
Tras definirlo, vamos a repasar cuatro bases de datos que cumplen con este método de publicación. El concepto de información en abierto llega hasta el punto de que nos ofrecen la posibilidad de descargarnos la base de datos al completo, además de consultarla online:
La misión de Uniprot es proveer a la comunidad científica con una herramienta sobre las secuencias de proteínas con información funcional, accesible, de alta calidad y libre. Familiarizarse con esta herramienta es muy sencillo gracias a sus tutoriales en vídeo.
Uniprot significa “Universal Protein Resource”, y se compone de las siguientes bases de datos:
● “UniProt Knowledgebase” (UniProtKB): Esta interfaz nos permite buscar en una colección de información funcional sobre proteínas con abundantes anotaciones. Además de ofrecer los campos clave de información: Secuencia de aminoácidos, nombre de la proteína y descripción, información taxonómica y citaciones), se añade información adicional como ontologías biológicas, clasificaciones y referencias cruzadas.
● “UniProt Reference Clusters” (UniRef): Ofrece conjuntos agrupados de secuencias de Uniprot KB, incluyendo las isoformas de las proteínas y registros seleccionados de UniParc para obtener una cobertura completa de la secuencia de sucesión en varias resoluciones ocultando secuencias redundantes (pero no su descripción).
● “UniProt Archive” (UniParc): Completa y no redundante base de datos que contiene la mayor parte de la información pública sobre secuencias de proteínas en el mundo.Es capaz de omitir las secuencias repetidas al extraerlas de diversas bases de datos.
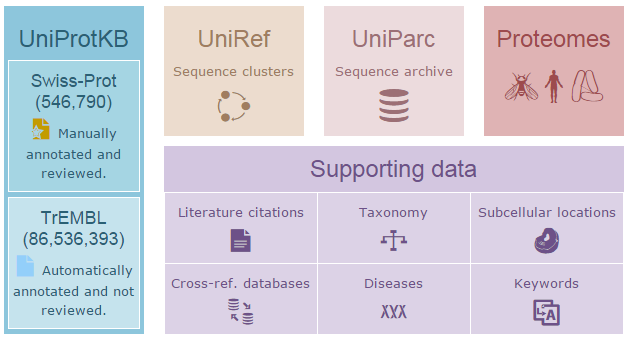
Fig. 2 Bases de datos y cifras de Uniprot
El proyecto Linked Clinical Trials (Linkedct) tiene por objetivo construir la primera fuente de información sobre ensayos clínicos en forma de web semántica abierta. La información que se genera en Linkedct se procesa de la siguiente manera:
1- Se transforma información existente sobre ensayos clínicos en RDF.
2- Se introducen enlaces entre los registros de la información de los ensayos clínicos y otras fuentes de información.
Fig. 3 Ensayos clínicos filtrados por “Estado”
Lanzado en el año 2004, ofrece información sobre la actividad biológica de las pequeñas moléculas. Está organizada como tres bases de datos enlazadas que son la siguientes: PubChem Substance, PubChem Compound, y PubChem BioAssay. También dispone de una herramienta de búsqueda rápida de similitudes químicas estructurales y un visor tanto 2D como 3D de las estructuras químicas.
Fig. 4 Visor 3D y comparador
Provee acceso a 25 bases de datos biomédicas desde un punto de acceso, lo que permite la consulta de 10 billones de resúmenes en RDF. Podemos buscar utilizando facetas como genes, proteínas, interacción molecular, objetivos, fármacos, ensayos clínicos y efectos secundarios. La interfaz es intuitiva y nos muestra sugerencias sobre lo que buscamos.
Fig. 5 Búsqueda por facetas
Imagen representación LOD: Linking Open Data cloud diagram 2014, by Max Schmachtenberg, Christian Bizer, Anja Jentzsch and Richard Cyganiak.
http://lod-cloud.net/